一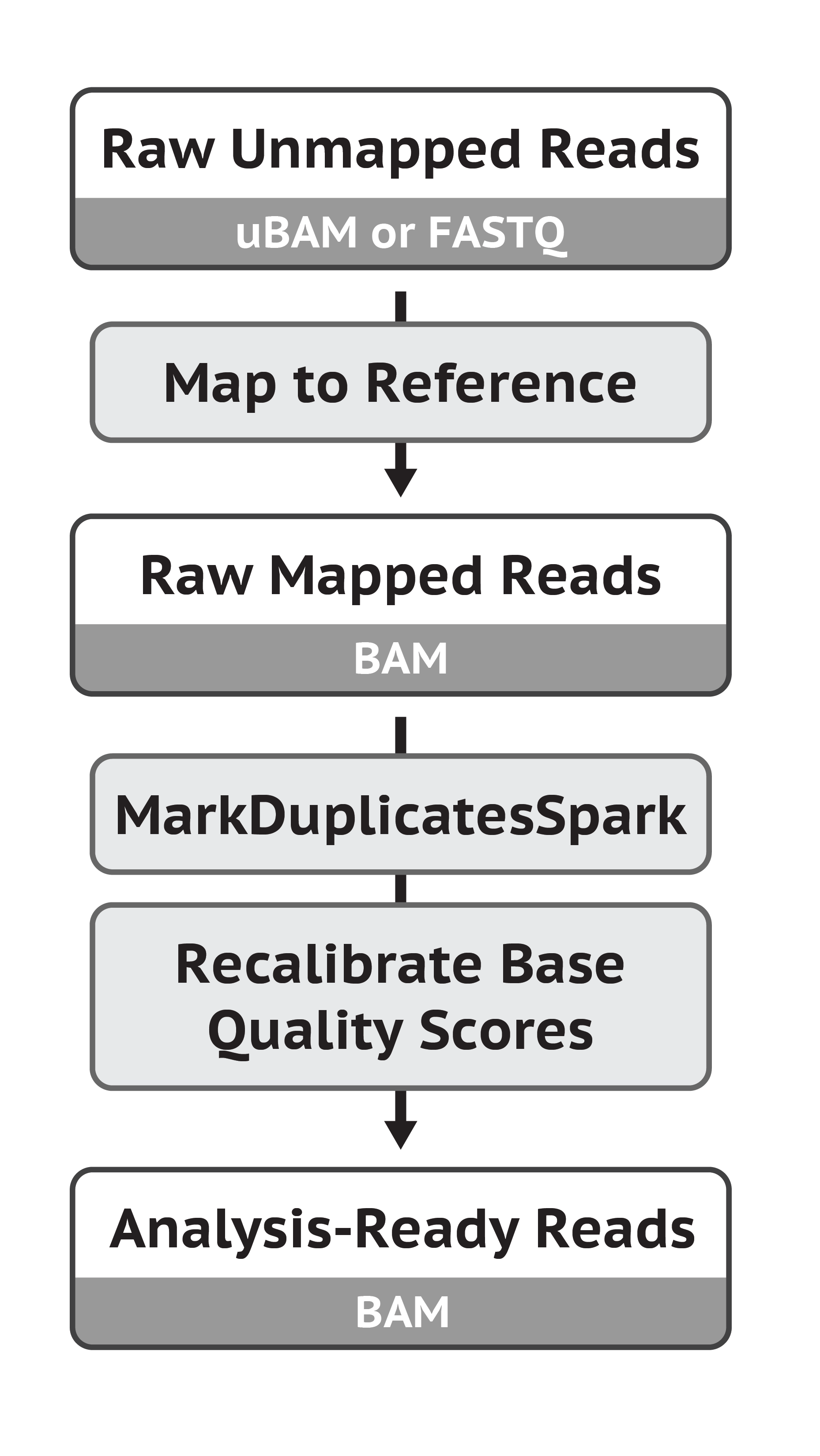
Pegasus是一个Workflow Management System。
利用它,设计一个DAG图作为一连串任务的流程,方便的进行并行、串行,并可从错误中恢复。
二、更多信息
https://pegasus.isi.edu/about/
三、GATK
illumina NGS的原理 https://blog.csdn.net/u010608296/article/details/88831797
Gatk Best Practice:https://software.broadinstitute.org/gatk/best-practices/workflow?id=11165
① 将Reads比对到参考基因组上:BWA
原理简述:
主要为了在内存消耗低的情况下实现序列比对(自然,动态规划是不行的)
Burrows-Wheeler数据压缩:
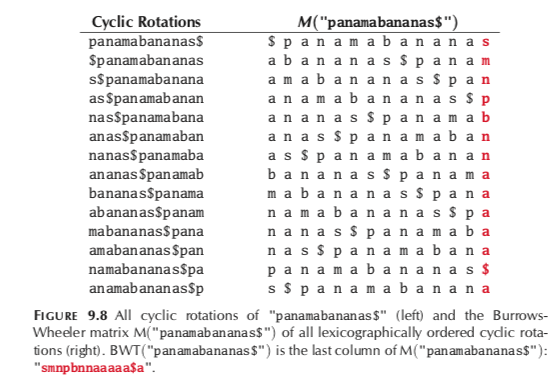
一种很自然的压缩方法是:ABAABADBADAD => 6A3B3D, 但是DNA序列不能这么压缩,因为DNA序列的信息蕴含在了碱基之间的顺序中。
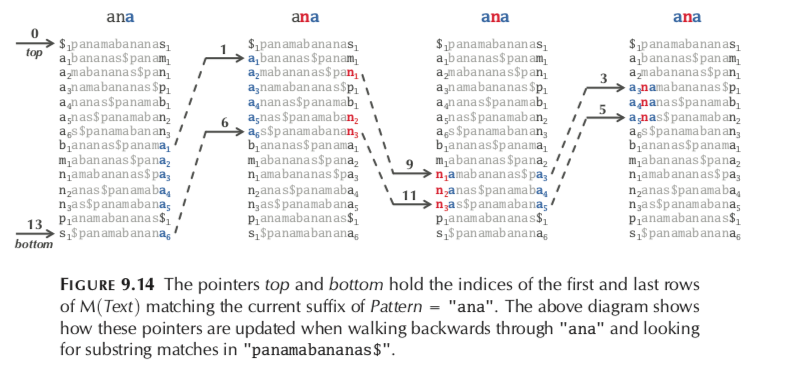
现在将序列“panamabananas”做如图的Burrows-Wheeler Transform,获得的矩阵按照首字母排序,可以发现后一列在原始字串中是位于列之前。可以看到,后一列有多个a连续出现,这是因为,在原始序列中 =,‘an’这个2-mer多次出现(在DNA序列中,这种情况很多),因此,我们对后一列进行压缩:SMNPB2N5A$A可以起到压缩作用(注意,ATCG组成的超长DNA中,这种压缩效果更显著)。由后一列,经过首字母排序,可以推导列。因此,在比对时,我们只用保存参考基因组的Burrows-Wheeler Matrix 的后一列的压缩形式。
右图:由于英文文章中,‘and’的出现次数很多,所以他的BWT变换后一列会出现连续的a(n、d亦然)。
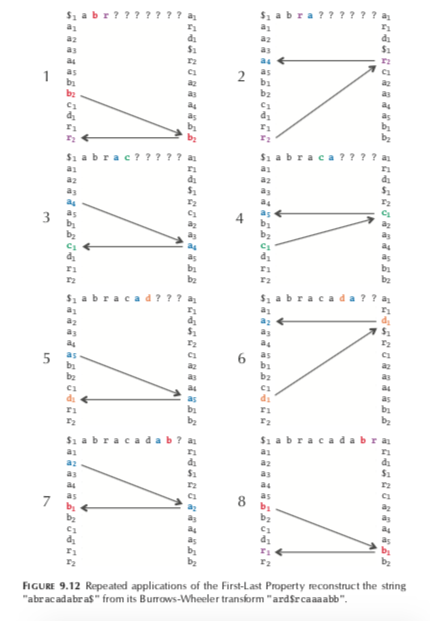
通过列与后一列,我们可以推导出整一个序列:后一列在原始字串中是位于列之前,所以,从后一列的$开始,看列的字符,然后再于后一列找刚才的字符,再看列。。。

 „
„
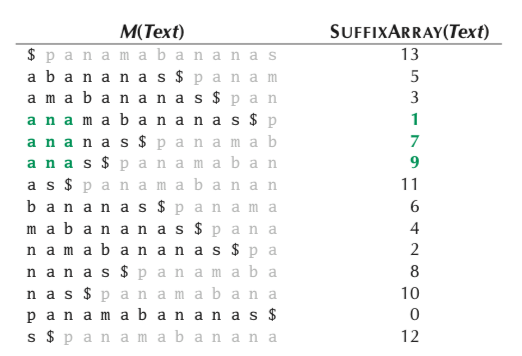
在将read比对到参考基因组上时,我们从read的后一位于BWMatrix后一列开始寻找,结果如右图所示:在开始的时候,我们为每一行算出一个SuffixArray(Text)作为定位参考。
现实中的比对是有错配的。在从列到后一列的对应搜寻中,就可以相应放宽要求。过程如下图,在次搜寻,引入一个错配,底下的情况,与第二次搜寻,又引入了一个错配,那么即停止并丢弃。
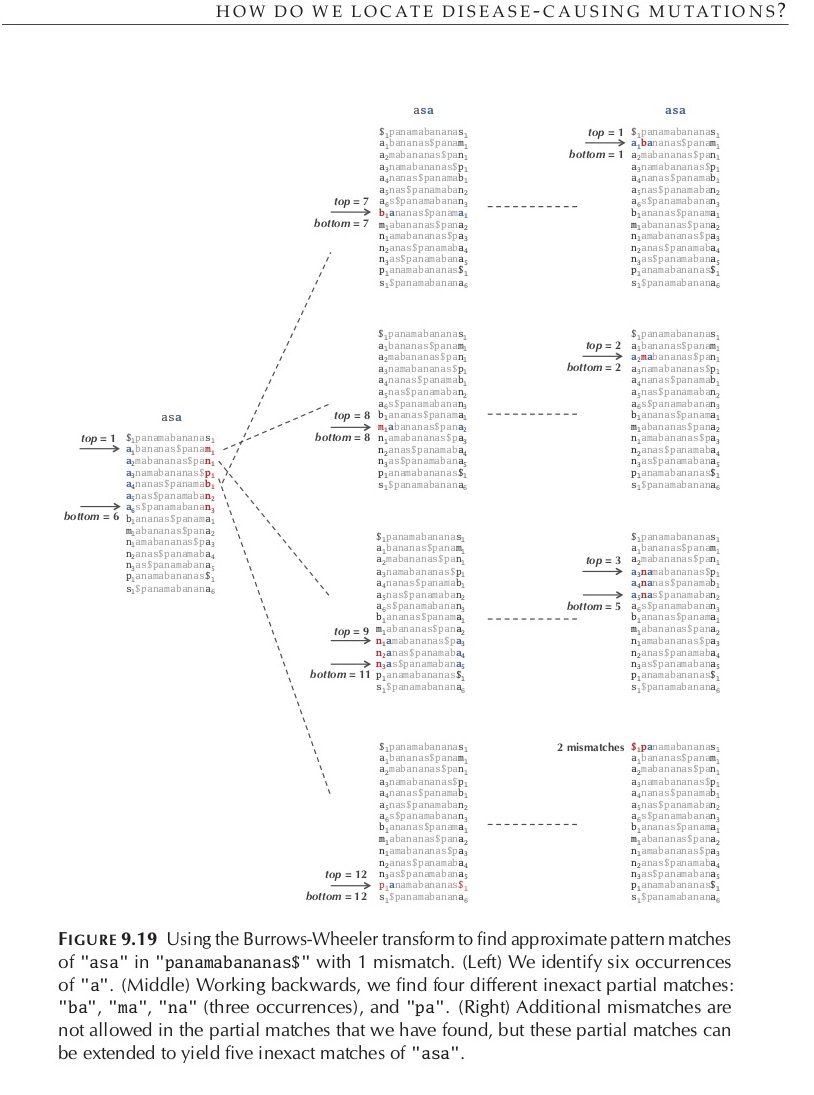
参看Phillip Compeau & Pavel Pevzner的Bioinformatics Algorithms: An Active Learning Approach
1 | bwa mem ref.fa read1.fq read2.fq > aln-pe.sam |

SN: Reference sequence name. The SN tags and all individual AN names in all @SQ lines must be distinct. The value of this field is used in the alignment records in RNAME and RNEXT fields. Regular expression: [:rname:∧*=][:rname:]*
LN: Reference sequence name. The SN tags and all individual AN names in all @SQ lines must be distinct. The value of this field is used in the alignment records in RNAME and RNEXT fields. Regular expression: [:rname:∧*=][:rname:]*
这步生成sam文件,然后要利用samtools将sam转为bam
SAM:Sequence Alignment Map
BAM:Binary Alignment Map
https://samtools.github.io/hts-specs/SAMv1.pdf
如果是将fastq文件分成多个来bwa的,现在要用picard将他们合并
1 | java -jar picard.jar MergeSamFiles <argument...> |
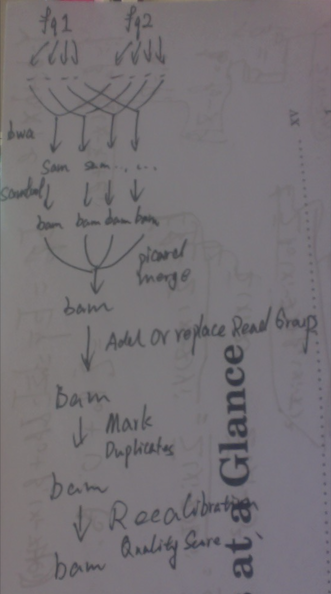
②Mark Duplicates:MarkDuplicatesSpark
- Reads are sorted into coorrdinate-order
- Mark the Duplicates, causing the marked pairs to be ignored by default during the variant discovery process.
它的意义参见 https://www.jianshu.com/p/cad6a359a368:SNP的发现
 View Code
View Code
❗️在这一步之前要加上头信息:利用picard的AddOrReplaceReadGroups
这个信息随便填也行,可以直接用示例
1 2 3 4 5 6 7 8 | java -jar picard.jar AddOrReplaceReadGroups \ I=input.bam \ O=output.bam \ RGID=4 \ RGLB=lib1 \ RGPL=ILLUMINA \ RGPU=unit1 \ RGSM=20 |
Mark Duplicates:
1 2 3 | gatk MarkDuplicatesSpark \ -I input.bam \ -O marked_duplicates.bam |
③ Base (Quality Score) Recalibration
- 这一步的目的是利用机器学习方法纠正由测序仪给出的质量分数(Base Quality Score)
- 这个误差来自:文库的制备,芯片生产,测序仪
- 校准方法:
- 收集covariate measurement from all base calls in datasets,建立统计模型。
- The initial statistics collection can be parallelized by scattering across genomic coordinates, typically by chromosome or batches of chromosomes but this can be broken down further to boost throughput if needed.
- Then the per-region statistics must be gathered into a single genome-wide model of covariation; this cannot be parallelized but it is computationally trivial, and therefore not a bottleneck.
- 利用这个模型对碱基质量进行校准 (可以并行)
- 收集covariate measurement from all base calls in datasets,建立统计模型。
四、利用Pegasus来跑GATK